ノーザンブロッティング
ノーザンブロッティング(英: Northern blotting、ノーザンブロット Northern blot とも)あるいはRNAブロッティング(英: RNA blotting、RNAブロット RNA blot とも)[1][2]とは、分子生物学研究において用いられる、RNAを検出することによって遺伝子の発現を解析する手法である[3][4]。「ノーザン(northern; 北の〜)ブロッティング」の名称は、DNAを検出するサザンブロッティングと同様の原理によることから、一種の洒落として名づけられた通称である[「サザンブロッティング」は開発者エドウィン・サザン(Southern; 英語で南の〜、の意)の名からとられている)][2][3]。

ノーザンブロッティングを用いることによって、形態形成や細胞分化、病気の発病などの様々な状況下で、ある特定の遺伝子がどのように発現しているかを比較・解析することが可能である[5]。ノーザンブロッティングにおいては、まずサンプルから抽出したRNAを電気泳動によってそのサイズごとに分離したのち、メンブレン(フィルター膜)への転写を行なう。その後RNAが転写されたメンブレン上で、標識した核酸プローブを用いて目的とするRNAの量、サイズを検出する。なお本来「ノーザンブロッティング」という言葉が指すのは、RNAを電気泳動ゲルからメンブレンへ転写(「ブロッティング」)する過程のみである。しかし、一般的には電気泳動、検出といったその前後の一連の過程を合わせて「ノーザンブロッティング」と呼ぶことが多い[2][6]。ノーザンブロッティングの手法は1977年にスタンフォード大学のJames Alwine、David Kemp、そしてGeorge Starkによって米国科学アカデミー紀要に発表された[7]。サザンブロッティングとの主な違いは、ノーザンブロッティングにおいてはDNAではなくRNAが解析の対象となることである[8]。
手法
概要
一般的なノーザンブロッティングの手順[6][9]は、まずホモジェナイズ(均質化)された組織または細胞サンプルからRNAを抽出することから始まる。真核生物のメッセンジャーRNA(mRNA)は、その末端にポリA尾部を持つことから、それに結合するオリゴdTを用いたクロマトグラフィーによって単離することができる[10][11]。こうして得られたRNAサンプルはゲル電気泳動によって分離される。電気泳動に用いるゲルは崩れやすく、またRNAの検出に用いる核酸プローブはゲルの基質に侵入することができないので、分離されたRNAは続いてメンブレンと呼ばれるナイロン膜へ、吸引もしくはキャピラリーを用いた拡散によって転写(ブロット)される。
ナイロンでできたメンブレンは正の電荷を帯びているため、負の電荷を帯びている核酸を効率よく吸着することができる。転写に用いるバッファーはホルムアミドを含んでおり、RNAとプローブのハイブリダイゼーションに必要な温度を下げる働きを果たしている。これにより、反応を低温で行うことができ、高温によるRNAの分解を防ぐことができる[12]。RNAがメンブレンに転写された後、UV照射や熱処理によって、RNAをメンブレンへ固定化する。その後、標識のついたプローブをRNAとハイブリダイズさせる。この段階でハイブリダイゼーションの効率と特異性に影響を与える因子として、イオン強度や粘度、RNAおよびプローブの長さ、不対合塩基対の数、塩基組成などが挙げられる[13]。ハイブリダイゼーションののちメンブレンを洗浄して、非特異的な結合を生じているプローブを洗い流す。ハイブリダイゼーションしたプローブとRNAの複合体を、標識の種類によってX線写真などの方法によって検出したのち、デンシトメトリーによってそのシグナルの強さを定量し、RNAの量を定量する。対照実験(ネガティブコントロール)として、マイクロアレイやRT-PCRによって標的のRNAが存在しないことが確認されているサンプルを用いることもある[13]。
ゲル
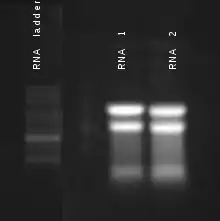
RNAサンプルはRNAの二次構造形成を阻害するための変性剤としてホルムアルデヒドを含んだアガロースゲルを用いた電気泳動によって分離されるのが一般的である[13][14]。泳動後のゲルをブロッティングする前にエチジウムブロマイドで染色し、UV照射下で観察することで、RNAの量と品質を確認することもある[13]。尿素を加えたポリアクリルアミドゲルを用いた電気泳動でもRNAを分離することができるが、これは断片化したRNAやマイクロRNAに対して用いられることが多い[15]。 サンプルの隣にはRNAラダー(分子量マーカー)を共に泳動して、得られたRNAの長さなどを調べることも多いが、全RNAサンプルの場合はリボソームRNAをマーカーの代わりとして用いることもできる[13]。リボソームの大サブユニットは分子量28S(約5,000 塩基対)、小サブユニットは分子量18S(約2,000 塩基対)であるため、泳動像には2本の目立つバンドが現れる(右図)[13][16]。
プローブ
ノーザンブロッティングにおけるRNAの検出には、標的RNAの一部または全部と相補的な塩基配列の核酸でできたプローブを用いる。プローブはDNAでもRNAでもよく、標的RNAと相補的な最低でも25塩基対以上のオリゴヌクレオチドであれば用いることができる[6]。一般に、in vitroでの転写反応によって作成されたRNAプローブの方が、複数回の洗浄に耐え、バックグランドのノイズを一部低減することができる[13]。プローブは放射性同位体(32P)、またはアルカリホスファターゼや西洋ワサビペルオキシダーゼ(HRP)などを用いた化学発光による方法で標識されるため、前者であれば写真の感光、後者であれば発光というかたちでプローブの存在する位置(すなわち、標的RNAの存在する位置)を検出できる[17]。化学発光による標識の方法は主に2つに分けられる。つまり、プローブを化学発光を起こす酵素で直接標識して検出する方法と、プローブを何らかの小分子(例えばビオチン)で標識し、それと結合する別の分子(例えばアビジンかストレプトアジビン)に化学発光を起こす酵素(例えばHRP)を繋げてプローブと結合させることで化学発光を起こす方法である[13]。放射性同位体による標識に比べ、化学発光による標識は短時間で検出が可能で感度も高い。また、前者は放射性物質を扱う事による健康への危険も伴うので、近年では放射性同位体を用いた方法よりも化学発光を用いた方法を用いる研究者が多い[17]。一枚のメンブレンで最大で5回、異なるプローブによる異なる標的遺伝子の検出を行うことができることが分かっている[12]。
応用
ノーザンブロッティングによって異なる組織、器官、環境ストレスのレベル、または感染の有無、何らかの処理の有無などによってある特定の遺伝子の発現がどのように変動しているかを解析することができる[11][18][19]。例えば、がん細胞において、通常の細胞と比べてがん遺伝子が過剰発現されていて、逆にがん抑制遺伝子の発現は下がっていることを示すのに、ノーザンブロッティングが用いられてきた[13]。他の例として、移植された臓器の拒絶反応における遺伝子の発現変化を調べるのに用いられたこともある[20]。ある特定の条件における遺伝子発現のパターンから、その遺伝子の機能を推測することもできる。RNAはまずそのサイズによって分離されるが、一つのプローブを用いてサイズの異なる複数のRNAが検出されれば、同一遺伝子における選択的スプライシングの存在、または共通する配列モチーフを持つ複数の遺伝子の存在などが示唆されることになる[10][16]。突然変異体における遺伝子の欠失や転写後修飾の異常によってRNAサイズの違いが生じる場合もあるが、これもノーザンブロッティングによって検出が可能である。同一遺伝子の中でも異なる標的配列に対して複数のプローブを設計することで、変異体においてRNAのどの部分で欠失が起こっているかを調べることもできる[3]。
利点と欠点
ノーザンブロッティングと同様に遺伝子発現を解析する手法として、RT-PCR、ヌクレアーゼプロテクションアッセイ、DNAマイクロアレイ、RNA-seq、SAGE法などが挙げられ、それぞれが場面に応じて使い分けられている[5][6]。例えばそのうちマイクロアレイを例にとると、この手法は通常1から少数の遺伝子のみを対象にするノーザンブロッティングと比べ、何千もの遺伝子の発現を同時に可視化できる点が優れている[19][21]。逆にノーザンブロッティングは、マイクロアレイでは検出できないような遺伝子発現における微小な差異を検出できることがある[21]。
RT-PCRと比較すると、ノーザンブロッティングは感度が低いという欠点があるものの、特異性が非常に高いため、偽陽性シグナルを検出してしまうことが少ないという利点がある[13]。その他、上述のようにRNAのサイズを検出し選択的スプライシングなどについても解析できる点や、メンブレンが数年単位で保管可能であるため追実験が可能である点などがノーザンブロッティングの利点として挙げられることがある[13]。
ノーザンブロッティングを行う際に頻発するトラブルとして、サンプルに残存していた、または環境中から混入したリボヌクレアーゼ(RNase)によってRNAサンプルが分解されてしまうことが挙げられる。これは使用器具を適切に滅菌すること、ジエチルピロカーボネート(DEPC)といったRNase阻害剤を用いることなどによって予防できる[6]。また、ノーザンブロッティングで用いられるDEPC、エチジウムブロマイド、放射性同位体といった薬品は、人体にとって危険なものが多いことにも注意が必要である[13]。
関連項目
出典
- Gilbert, S. F. (2000) Developmental Biology, 6th Ed. Sunderland MA, Sinauer Associates.
- “RNAブロット法”. 光合成事典. 日本光合成学会. 2020年9月21日閲覧。
- Alberts, B., Johnson, A., Lewis, J. Raff, M., Roberts, K., Walter, P. 2008. Molecular Biology of the Cell, 5th ed. Garland Science, Taylor & Francis Group, NY, pp 538–539.
- Kevil, C. G., Walsh, L., Laroux, F. S., Kalogeris, T., Grisham, M. B., Alexander, J. S. (1997) An Improved, Rapid Northern Protocol. Biochem. and Biophys. Research Comm. 238:277–279.
- Schlamp, K.; Weinmann, A.; Krupp, M.; Maass, T.; Galle, P. R.; Teufel, A. (2008). “BlotBase: A northern blot database”. Gene 427 (1–2): 47–50. doi:10.1016/j.gene.2008.08.026. PMID 18838116.
- Trayhurn, P. (1996) Northern Blotting. Pro. Nutrition Soc. 55:583–589.
- “Method for detection of specific RNAs in agarose gels by transfer to diazobenzyloxymethyl-paper and hybridization with DNA probes”. Proc. Natl. Acad. Sci. U.S.A. 74 (12): 5350–4. (1977). doi:10.1073/pnas.74.12.5350. PMC 431715. PMID 414220.
- Bor, Y.C.; Swartz, J.; Li, Y.; Coyle, J.; Rekosh, D.; Hammarskjold, Marie-Louise (2006). “Northern Blot analysis of mRNA from mammalian polyribosomes”. Nature Protocols. doi:10.1038/nprot.2006.216
- “蛍光検出基礎知識 検出の達人への道⑤ 核酸のブロッティングと標識・検出”. 蛍光アプリケーション~milestone. GEヘルスケアバイオサイエンス (2004年12月). 2020年9月21日閲覧。
- Durand, G. M.; Zukin, R. S. (1993). “Developmental Regulation of mRNAs Encoding Rat Brain Kainate/AMPA Receptors: A Northern Analysis Study”. J. Neurochem. 61 (6): 2239–2246. doi:10.1111/j.1471-4159.1993.tb07465.x. PMID 8245974.
- Mori, H.; Takeda-Yoshikawa, Y.; Hara-Nishimura, I.; Nishimura, M. (1991). “Pumpkin malate synthase Cloning and sequencing of the cDNA and Northern blot analysis”. Eur. J. Biochem. 197 (2): 331–336. doi:10.1111/j.1432-1033.1991.tb15915.x. PMID 1709098.
- Yang, H.; McLeese, J.; Weisbart, M.; Dionne, J.-L.; Lemaire, I.; Aubin, R. A. (1993). “Simplified high throughput protocol for Northern hybridization”. Nucleic Acids Research 21 (14): 3337–3338. doi:10.1093/nar/21.14.3337. PMC 309787. PMID 8341618.
- Streit, S.; Michalski, C. W.; Erkan, M.; Kleef, J.; Friess, H. (2009). “Northern blot analysis for detection of RNA in pancreatic cancer cells and tissues”. Nature Protocols 4 (1): 37–43. doi:10.1038/nprot.2008.216. PMID 19131955.
- Yamanaka, S.; Poksay, K. S.; Arnold, K. S.; Innerarity, T. L. (1997). “A novel translational repressor mRNA is edited extensively in livers containing tumors caused by the transgene expression of the apoB mRNA-editing enzyme”. Genes Dev. 11 (3): 321–333. doi:10.1101/gad.11.3.321. PMID 9030685.
- Valoczi, A., Hornyik, C., Varga, N., Burgyan, J., Kauppinen, S., Havelda, Z. (2004) Sensitive and specific detection of microRNAs by northern blot analysis using LNA-modified oligonucleotide probes. Nuc. Acids Research. 32: e175.
- Gortner, G.; Pfenninger, M.; Kahl, G.; Weising, K. (1996). “Northern blot analysis of simple repetitive sequence transcription in plants”. Electrophoresis 17 (7): 1183–1189. doi:10.1002/elps.1150170702. PMID 8855401.
- Engler-Blum, G.; Meier, M.; Frank, J.; Muller, G. A. (1993). “Reduction of Background Problems in Nonradioactive Northern and Southern Blot Analysis Enables Higher Sensitivity Than 32P-Based Hybridizations”. Anal. Biochem. 210 (2): 235–244. doi:10.1006/abio.1993.1189. PMID 7685563.
- Liang, P. Pardee, A. B. (1995) Recent advances in differential display. Current Opinion Immunol. 7: 274–280.
- Baldwin, D., Crane, V., Rice, D. (1999) A comparison of gel-based, nylon filter and microarray techniques to detect differential RNA expression in plants. Current Opinion in Plant Biol. 2: 96–103.
- Utans, U.; Liang, P.; Wyner, L. R.; Karnovsky, M. J.; Russel, M. E. (1994). “Chronic cardiac rejection: Identification of five upregulated genes in transplanted hearts by differential mRNA display”. Proc. Natl. Acad. Sci. USA 91 (14): 6463–6467. doi:10.1073/pnas.91.14.6463. PMC 44222. PMID 8022806.
- Taniguchi, M.; Miura, K.; Iwao, H.; Yamanaka, S. (2001). “Quantitative Assessment of DNA Microarrays – Comparison with Northern Blot Analysis”. Genomics 71 (1): 34–39. doi:10.1006/geno.2000.6427. PMID 11161795.