シロイヌナズナ
シロイヌナズナ(白犬薺、学名:Arabidopsis thaliana)は、アブラナ科シロイヌナズナ属の一年草。植物のモデル生物として有名。
| シロイヌナズナ | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 シロイヌナズナ | ||||||||||||||||||||||||
| 分類 | ||||||||||||||||||||||||
| ||||||||||||||||||||||||
| 学名 | ||||||||||||||||||||||||
| Arabidopsis thaliana | ||||||||||||||||||||||||
| シノニム | ||||||||||||||||||||||||
|
Arabis thaliana[1] | ||||||||||||||||||||||||
| 和名 | ||||||||||||||||||||||||
| シロイヌナズナ(白犬薺) | ||||||||||||||||||||||||
| 英名 | ||||||||||||||||||||||||
| Thale Cress, Mouse-ear Cress | ||||||||||||||||||||||||
 シロイヌナズナの生息域 |

概要
ユーラシア大陸からアフリカ大陸北部原産の越年草である[2][3][4][5][6][7][8]。ヨーロッパ、アジア、アフリカに自生し、地理的には地中海からスカンジナビア、スペインからギリシャまでほぼ連続的に分布する[9]。また、熱帯高山気候のアフリカや南アフリカにも自生している[10][11]。17世紀頃に北アメリカなど世界各地で導入・帰化されており[12].[13]、日本においては帰化植物として、北海道から九州の海岸や低地の草地に分布する。岩場、砂地、石灰質といった土壌でも容易に生育することができ、先駆植物となることもある。農耕地、道端、線路、荒地など攪乱された場所に広く分布するため、一般に雑草とみなされる[12][14]が、競争力が低く、背丈も小さいため、有害雑草には分類されない[15]。他のアブラナ科の種と同様に、食用にすることもできるが、野菜や山菜として広く利用されることはない[16]。
生活型はロゼット型で、花茎は高さ10-30cmに達する[7]。長日植物で花期は4-6月。茎頂に総状花序を出し、花は白色で花弁を4枚持ち、多数つく。大抵は自家受粉によって種子をつくり、20~30個の種子を含む長角果の果実をつける[17][18][19][20]が、稀にハナバチやアザミウマなどといった昆虫による他家生殖も行う[21]。葉や茎には毛があり、ロゼット葉は2-5cmほど、茎葉は2cm前後の長楕円形である。根は単純な構造になっており、一本の主根が垂直に下に伸びていき、後に小さな側根を生じる。この根はBacillus megateriumなどの根圏細菌と相互作用する[22]。発芽から種が付くまで1〜2か月と短い[2]。実験室では、シャーレやプラントポットを使ったり水耕栽培をしたりすることで育てられ、蛍光灯の光でも温室でも育てることができる[23]。
ゲノムサイズが小さいこと、一世代が約2ヶ月と短いこと、室内で容易に栽培できること、多数の種子がとれること、自家不和合性を持たないこと、形質転換が容易であることなど、モデル生物としての利点を多く備えているため、研究材料として利用される。また、シロイヌナズナは、自生していた地域や、後年に導入された地域などから数千の天然近交系接合が収集されており[24]、遺伝背景や表現型に多彩なバリエーションを持つ個体群が収集されているため、シロイヌナズナは異なる環境への適応を研究するために用いることができる[24]。多くの変異系統が維持されており、日本国内では理化学研究所バイオリソースセンターやかずさDNA研究所などで、cDNA情報の公開、変異株の収集・維持・配布を行っている。
発見と名前の由来
シロイヌナズナは1577年に、ドイツテューリンゲン州ノルトハウゼンの医師ヨハネス・タール(Johannes Thal、1542年 - 1583年)によってハルツ山地で初めて発見され、Pilosella siliquosaと命名された。1753年にカール・フォン・リンネによって、発見者であるタールに因んでArabis thalianaと改名され、1842年に、ドイツ人植物学者グスタフ・ハインホルトによって新しい属Arabidopsis属(シロイヌナズナ属)が提唱された際に、その中に位置づけられた。属名のArabidopsisは、ギリシア語で「Arabisに似ている」を意味する。
モデル生物としてのシロイヌナズナ
シロイヌナズナの研究は、1900年代初頭に生物学者や植物学者によって始められ、1945年頃に初めてシロイヌナズナの突然変異体に関する系統的な記述がされた[25]。現在、シロイヌナズナは、集団遺伝学を含む遺伝学、植物の進化や発生などに関する植物科学の研究に広く用いられている[26][27][28]。シロイヌナズナ自体には農業において直接的な意義はほとんどないが、いくつかの特徴を備えていることから花き植物の遺伝、細胞、分子生物学を理解するのに良いモデルとして知られている。

1873年、アレクサンダー・ブラウンによって、シロイヌナズナの最初の突然変異体が報告され、ここで八重咲きという表現型が示された(変異遺伝子は1990年にクローニングされ、Agamousとされた)[29]。ドイツの植物学者フリードリヒ・ライバッハは、1907年に染色体数を発表したものの、1943年までシロイヌナズナをモデル生物として提案しなかった[30]。彼の教え子であるエルナ・ラインホルツは、1945年に執筆した論文の中で、X線突然変異誘発法を用いて作成した最初の突然変異体コレクションについて発表した[31][30]。ライバッハはその後も、多数の「エコタイプ」と呼ばれるような変異体を収集し、シロイヌナズナの研究に重要な貢献をした。Albert Kranzの協力により、これらは世界中の750の自然接種からなる大規模なコレクションとして整理された。
1950年代から1960年代にかけて、John LangridgeとGeorge Rédeiらの活躍により、シロイヌナズナは生物学的実験に有用な生物として確立されていった。Rédeiは、このモデルを科学界に紹介するために総説論文を複数発表した。1964年にはArabidopsis Information Serviceというニューズレターが設立された[32]ことで、シロイヌナズナの研究コミュニティが始動し、1965年には、ドイツのゲッティンゲンで最初の国際会議が開催された。
1980年代に入り、シロイヌナズナは世界中の植物研究室で広く利用されてくるようになった。他に植物の研究に用いられる候補として、既に植物生物学の遺伝的モデルとして確立されていたトウモロコシや、当時の技術で容易に形質転換が可能であったペチュニア、タバコなどがあった[30]。その中で、シロイヌナズナがモデル植物として特に広く使われるようになるきっかけの年となったのは、シロイヌナズナでのT-DNAを介した形質転換と、初めてクローニングされたシロイヌナズナ遺伝子が報告された1986年であった[33][34]。
ゲノム
核ゲノム
2000年12月に植物としては初めて全ゲノム解読が終了した[37]。シロイヌナズナはゲノムサイズが小さく、2倍体であるため、遺伝子マッピングや配列決定に有用である。ゲノムサイズはおよそ1.57億塩基対[38]、染色体は5対、遺伝子数は約2万7600個と顕花植物では最小の部類に入る[39]。長い間、シロイヌナズナは顕花植物の中で最も小さなゲノムを持っていると考えられていたが、2014年にシソ目タヌキモ科ゲンリセア属の食虫植物であるGenlisea tuberosaがシロイヌナズナより小さい約61Mbpのゲノムを持つことが発見された[40]。
このゲノムには、約2万7600個のタンパク質コード遺伝子と約6500個のノンコーディングRNA遺伝子が含まれている[41]が、Uniprotデータベースには、シロイヌナズナのリファレンスプロテオームとして39,342個のタンパク質が掲載されている[42]。2017年時点で、この約2万7600個のタンパク質コード遺伝子のうち、2万5402個(およそ91.8%)にはタンパク質名が推測され、割り振られている[43]が、これらのタンパク質の中には、その性質や役割が十分理解されておらず、名前だけが一般的に知られているもの(例えば「特異性の分からないDNA結合タンパク質」のようなもの)であると考えられる部分が多く存在している。また、Uniprotでは、リファレンスプロテオームの一部として、3,000以上のタンパク質を「uncharacterized」としてリストアップしている。
葉緑体ゲノム
シロイヌナズナの葉緑体ゲノムの長さは154,478塩基対で[35]、これは多くの顕花植物で典型的に見られるサイズである。小サブユニットリボソームタンパク質(rps、黄色:右図参照)、大サブユニットリボソームタンパク質(rpl、オレンジ)、オープンリーディングフレームではないかとされるタンパク質(ycf、レモン)、光合成に関連するタンパク質(緑)、その他の機能に関連するタンパク質(赤)、リボソームRNA(rrn、青)および転移RNA(trn、黒)をコードする136遺伝子から構成されている[36]。
ミトコンドリアゲノム
シロイヌナズナのミトコンドリアゲノムは367,808塩基対の長さで、57の遺伝子を含んでいる[44]。シロイヌナズナのミトコンドリアゲノムには、多くの繰り返し領域が含まれており、最も大きな繰り返しは定期的に組み換えられ、ゲノムを異性化する[45]。多くの植物におけるミトコンドリアゲノムと同様に、シロイヌナズナのミトコンドリアゲノムも生体内では分岐分子と直鎖分子が重なり合った複雑な配列として存在している[46]。
遺伝学
シロイヌナズナの遺伝子組み換えに際しては、植物に対して病原性を持つアグロバクテリウムと呼ばれる土壌細菌(Rhizobium radiobacter[注釈 1])を利用して植物ゲノムにDNAを導入する方法が広く用いられている。2022年現在、主流となっているプロトコルは「フローラルディップ法」と呼ばれ、目的のプラスミドを持つアグロバクテリウムと界面活性剤を含む溶液に花を浸すだけで行うことができる方法[47][48]であり、この方法では、組織培養や植物体の再生といった操作は必要ない。
シロイヌナズナの遺伝子ノックアウトコレクションは、ゲノミクスリソースに対する資金提供と高効率な形質転換の技術によって可能となった、植物科学において他に類を見ないリソースである。T-DNAの挿入部位が異なる30万以上の独立したトランスジェニック系統があり、このトランスジェニック系統に関する情報と種子はオンラインのT-DNAデータベースからアクセス可能である[49]。このコレクションがあることによって、研究者たちはシロイヌナズナが持つほとんどの遺伝子についての挿入型変異体を手に入れることができる。
シロイヌナズナの各種変異系統や特徴的な系統は、実験室での研究において実験材料として利用される。最もよく使われる背景系統にはLer(Landsberg erecta)およびCol(Columbia)がある[50]。科学論文にあまり引用されない背景系統としては、Ws(Wassilewskija)、C24、Cvi(Cape Verde Islands)、Nossenなどがある[51]。このような系統と近縁のCol-0やCol-1などと命名されたアクセッションが得られ、その特性が評価されており、一般的に、これらの変異株はストックセンターを通じて入手可能である。中でもNottingham Arabidopsis Stock Center-NASC[50]やアメリカ・オハイオ州のArabidopsis Biological Resource Center-ABRC は有名である[52]。Col-0は、RédeiがLaibachから受け取った「Landsberg」と名付けられた放射線照射されていない種子集団から選抜された[53]。Columbia(Rédeiの前任地であるミズーリ大学コロンビア校の所在地から命名された)は、Arabidopsis Genome Initiativeで配列決定したリファレンスアクセッションである[37]。Later(Landsberg erecta)系統は、RédeiがX線で変異させたLandsberg集団から草丈の低いものを選抜した系統で、Lerの変異体コレクションは、このX線で変異させた系統に由来するため、Ler-0は、La-0やLa-1などを含むLandsbergのアクセッションには対応していない。
毛状突起(トライコーム)の形成はGLABROUS1タンパク質によって開始される。このタンパク質に対応する遺伝子をノックアウトすると、無毛の植物が得られる。この表現型はすでにゲノム編集の実験に利用されており、CRISPR/Cas9などのゲノム編集技術を改良するための植物研究における視覚的なマーカーとして注目されるかもしれない[54][55]。
非メンデル性遺伝に関する論争
2005年、パデュー大学の研究者たちは、シロイヌナズナが既知のDNA修復のメカニズムに代わるものを持っており、異常な遺伝パターンを生み出すと提唱したが、観察された現象(HOTHEAD遺伝子の変異体のコピーが野生型に戻る現象)は、後に人工的なものであると示唆された[56][57][58]。
ライフサイクル
サイズが小さく、ライフサイクルが早い植物であることも、研究に使う上で有利である。シロイヌナズナは、発芽から種子の成熟まで約6週間しかかからないため、短期間で実験に使える系統を発見するのに使われてきた。また、植物体が小さいため、狭い場所で栽培する際に便利であり、多くの種子を生産することができる。さらに、この植物が持つ自殖性は、遺伝子実験に役立つ。また、1株で数千個の種子を生産することができる。このように多くの利点があることから、シロイヌナズナはモデル生物として評価されるに至った。
細胞生物学
植物におけるSNAREタンパク質の研究では、シロイヌナズナをモデルとして用いることが多い。これにより、SNAREが小胞輸送に大きく関与していることが明らかになった。Zhengらは、1999年にAtVTI1aと呼ばれるシロイヌナズナのSNAREがゴルジ体 - 液胞間の物質輸送に必須である可能性を見出した。これはまだ未解決の分野であり、植物のSNAREが物質輸送に果たす役割は、まだ十分に解明されていない[59]。
DNA修復
植物のDNAは紫外線に弱く、紫外線によるゲノム損傷を回避したり修復したりするために、DNA修復機構が進化している。紫外線による代表的なDNA損傷としてシクロブタンピリミジン二量体(CPD)の生成があげられる。DNAを構成する4種類の塩基のうち、シトシンやチミンは、分子構造中にピリミジン骨格を持っているが、この骨格は紫外線を吸収して光化学反応を起こす。同じDNA一本鎖上でピリミジン骨格を持つ塩基が連続して2つある場合、共有結合によってシクロブタン構造を形成することで二量体であるCPDになる。このCPDが生成されると、DNAの転写や複製の妨げとなり、細胞に様々な弊害をもたらすことが知られている[60]。Kaiserらは、シロイヌナズナにおいて、CPDとなってしまった塩基が、CPDフォトリアーゼの発現によって修復されることを示した[61]。
宇宙での生育
1982年、ソ連の宇宙ステーションであるサリュート7号の乗組員がシロイヌナズナを栽培し、宇宙で花を咲かせて種子を作った最初の植物となったが、給水設備の不調により、2008年6月、スペースシャトル・ディスカバリーにて地球に持ち帰られることになった。この時のシロイヌナズナのライフスパンは、40日間であった[62]。2019年には、学生実験の一環として、中国の月探査船・嫦娥4号によってシロイヌナズナの種子が月へ運ばれた[63]。2022年5月12日、NASAは、シロイヌナズナの標本を月のレゴリスのサンプルで発芽・生育させることに成功したと発表した[64]。 発芽・育苗に成功したものの、対照群として火山灰で育てた標本ほど健康には育たなかった。また、アポロ12号やアポロ17号で採取したレゴリスで育てたシロイヌナズナはアポロ11号で採取したレゴリスで育てたものより健康だったことから、レゴリスの採取場所によって植物の生育に違いがあることも分かった[65]。
また、欧州宇宙機関により、国際宇宙ステーションでシロイヌナズナの継続的な研究が行われている。その目的は、微小重力下で植物の種から種への成長と繁殖を研究することにある[66][67]。
発生

花器官の形成
シロイヌナズナは、花の形成のモデルとして広く研究されている。花は、萼(がく)片、花弁、雄蕊(おしべ)、心皮(めしべの構成単位)という4つの基本的な器官から構成されている。これらの器官は、外側に4枚の萼片、その内側に4枚の花弁、6本の雄蕊、そして中央の心皮という順に、同心円に配列されている。シロイヌナズナのホメオティック変異では、ある器官が別の器官に変わってしまう。例えば、agamous変異の場合、雄蕊は花弁になり、心皮は萼片に置き換えられ、更にその内側に花弁と萼片のみからなる二次花が形成されることで、萼片 - 花弁 - 花弁というパターンが再帰的に繰り返されることになり、結果として八重咲きの表現型を示す。
このようなホメオティック変異の観察から、E. CoenとE. Meyerowitzは花の発生に関するABCモデルを定式化した[68]。
このモデルによると、花器官の形成を制御する遺伝子は、クラスA遺伝子(がく片と花弁に影響)、クラスB遺伝子(花弁とおしべに影響)、クラスC遺伝子(おしべとめしべに影響)の3クラスに分類される。これらの遺伝子は転写因子をコードしており、発生過程でそれぞれの組織を指定するために組み合わされる。このモデルはシロイヌナズナの花の研究を通して発見されたが、他の顕花植物にも一般に適用することができる。
葉の形成
シロイヌナズナを用いた研究は、特に双子葉植物における葉の形態形成の遺伝学に関して、重要な知見を与えている[69][70]。このような知見の多くは、葉の発達に関わる変異体の分析からもたらされたが、変異体のうちいくつかは1960年代に同定されていたものの、遺伝的および分子生物学的技術を使った分析は1990年代中頃まで行われていなかった。シロイヌナズナの葉は比較的単純で安定しているため、葉の発生の研究によく適している。
シロイヌナズナを用いた研究により、葉が形成される背景にある遺伝学がより明確に分かり、3つの段階に大別されるようになった。葉原基の形成、背腹性の確立、そして周縁分裂組織の形成である。葉原基の形成は、クラスI KNOXファミリー(SHOOT APICAL MERISTEMLESSなど)の遺伝子とタンパク質が抑制されることで開始する。これらのクラスI KNOXタンパク質は、葉原基におけるジベレリン生合成を直接的に抑制する。葉原基におけるこれらのクラスI KNOX遺伝子の抑制には、ASYMMETRIC LEAVES1、BLADE-ON-PETIOLE1、SAWTOOTH1など多くの遺伝的要因が関与していることがわかった。この抑制により、ジベレリン濃度が上昇し、葉の原基が成長を開始する[71]。
顕微鏡観察
シロイヌナズナは、光学顕微鏡による分析にも適している。シロイヌナズナの幼苗は全体的に、特に根は比較的半透明であるため、その小ささと相まって、蛍光顕微鏡や共焦点レーザー顕微鏡を用いて生細胞イメージングを行いやすい[72]。 水中や培地中で苗をマウントすることにより、固定や切片化することなく、非侵襲的にイメージングすることができ、タイムラプス測定も可能である[73]。蛍光タンパク質のコンストラクトを形質転換により導入することもできるため、これを用いれば植物体内の各種タンパク質の位置や蛍光タンパク質マーカーから各細胞の発生段階を推定することができるため、詳細な発生解析が可能である。
生理学
光センシング、光放射、時間生物学
光受容体であるフィトクロムA、B、C、D、Eは、赤色光に基づく光応答を媒介する。これらの受容体の機能に関する研究は、植物の光周性、発芽、脱落、避陰反応を制御するシグナル伝達カスケードを植物生理学者が理解するのに役立っている。FCA[74]、fy[74]、fpa[74]、LUMINIDEPENDENS(ld)[74]、fly[74]、fve[74]、FLOWERING LOCUS C(FLC)[75][76]といった遺伝子は、開花と春化に関する光周期のトリガーに関与していることが分かっている。Leeらは1994年にldがホメオドメインを生成することを、Blazquezらは2001年にfveがWD40リピートを生成することを発見している[74]。
UVR8タンパク質は、DNAに損傷を与えるUV-B光を検出し、それに対する応答を媒介する。
シロイヌナズナは、光屈性、葉緑体定位運動、気孔の開閉などの青色光に影響されるプロセスに関する遺伝的メカニズムの研究にも広く使用された[77]。これらの形質は、フォトトロピンという光受容体によって感知される青色光に応答している。シロイヌナズナは、もう一つの青色光受容体であるクリプトクロムの機能を理解する上でも重要であり、特に植物の概日リズムを制御するための光同調に重要である[78]。暗期の開始が異常に早いと、シロイヌナズナはデンプンの代謝を、細胞分裂に必要となる量まで低下させてしまう[79]。
光応答は、それまで光にほとんど反応しないと考えられていた根にも見いだされた。シロイヌナズナの根は、通常は重力屈性反応が優勢であるが、変異原で処理し、重力屈性がないことを選別したサンプルは、青色または白色光に負の、赤色光に正の光屈性反応を示し、根も正の光屈性を示すことが示された[80]。
2000年、アメリカ・ライス大学の植物生物学者Janet Braamは、遺伝子工学的手法により、触ると暗闇で光るシロイヌナズナを作製した。この効果は、超高感度カメラで見ることができた[81]。
「Glowing Plant」プロジェクトをはじめ、シロイヌナズナを使って、植物の発光強度を商業的に可能なレベルまで高めようとする試みが複数進められている。
植物病理学
植物が病害への抵抗性を獲得するメカニズムを理解することは、世界の農業と食糧生産を守るために重要である。植物と細菌、真菌、卵菌、ウイルス、線虫といった病原体との相互作用をよりよく理解するために、多くのモデル系が開発されてきており、シロイヌナズナは、植物病理学という学問分野、すなわち植物と病気を引き起こす病原体との相互作用の研究において、強力なツールとなっている[82]。
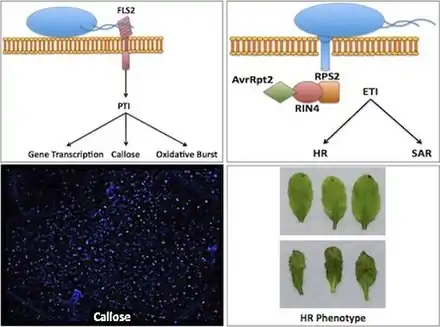
PAMPトリガー免疫の図解。具体的にはFLS2によるフラジェリン認識(左上)、RIN4を通したRPSによるavrRpt2の認識を通じて描かれるエフェクタートリガー免疫(右上)、シロイヌナズナの葉におけるカロース沈着の顕微鏡写真(左下)、シロイヌナズナにおける過敏感反応(HR)なしの(右下図中上)およびHRの様子(右下図中下)。
| 病原体の種類 | シロイヌナズナでの例 |
|---|---|
| 細菌類 | Pseudomonas syringae、Xanthomonas campestris |
| 真菌類 | Colletotrichum destructivum、Botrytis cinerea、Golovinomyces orontii |
| 卵菌類 | Hyaloperonospora arabidopsidis |
| ウイルス | カリフラワーモザイクウイルス(CaMV)、トマトモザイクウイルス(TMV) |
| 線虫 | Meloidogyne incognita、Heterodera schachtii |
シロイヌナズナを用いた研究は、植物がどのように病害への抵抗性を発現するかを調べるにあたって、多くのブレイクスルーをもたらした。多くの植物がほとんどの病原菌に対して抵抗性を示すのは、非宿主抵抗性によるもので、あらゆる病原菌がすべての植物に感染するわけではない。例えば、イネ科のうどんこ病の原因菌であるBlumeria graminisは、非宿主抵抗性の原因となる遺伝子を特定するためにシロイヌナズナが利用された代表例である。変異原であるメタンスルホン酸エチルを用いてシロイヌナズナの変異体を開発し、B. graminisによる感染が増加した変異体を同定した[84][85][86]。感染率が増加した変異体は、病原菌がシロイヌナズナに侵入(penetrate)して発病を開始できることから、PEN変異体と呼ばれるようになった。後に、B. graminisに対する非宿主抵抗性の原因となる遺伝子を特定する研究が進み、PEN遺伝子のマッピングが行われた。
一般に、植物が病原体や非病原性の微生物にさらされると、PAMPトリガー免疫(PAMP-triggered immunity, PTI)と呼ばれる初期反応が起こるが、これは植物がPAMPs(Patogen-Associated molecular patterns)と呼ばれる分子モチーフを検出するためである[87]。これらのPAMPsはある種の微生物においてよく保存されており、植物細胞表面のパターン認識受容体(Pattern recognition receptors, PRRs)と呼ばれる特殊な受容体で検出される。
シロイヌナズナのPRRのうち、最もよく知られているのはFLS2(Flagellin-Sensing2)で、これは細菌の鞭毛を構成するタンパク質の一種であるフラジェリンがN末端に持つ22個のアミノ酸からなるリガンド「flg22」を認識し、植物の防御システムを活性化する[88][89][90]。FLS2の発見は、flg22を認識することができないシロイヌナズナのエコタイプWs-0の同定により進められ、最終的にFLS2をコードする遺伝子の同定に至った。FLS2は、1995年に初めて単離されたPRRであるイネのXA21と同じく、インターロイキン1受容体関連キナーゼ(interleukin-1 receptor-associated kinase, IRAK)ファミリーと呼ばれるグループに属する[91]。Molinierらが2006年の論文で示した[92]ように、フラジェリンとUV-Cは、シロイヌナズナにおいて相同組換えを増加させるように作用しており、これは体細胞への影響のみにとどまらず、後続世代にも広がることが見出されている[93]。
シロイヌナズナで同定された第2のPRRであるEF-Tu receptor(EFR)は、タンパク質合成に用いられる原核生物の伸長因子である細菌のEF-Tuタンパク質や、それに由来するペプチドであるelf18をリガンドとして認識する[94]。Séverine Lacombeらは2010年に発表した論文の中で、アグロバクテリウムを用いた形質転換法により、元来EF-Tuを認識しない植物であるNicotiana benthamiana(ベンサミアナタバコ)にEFR遺伝子を導入し、細菌のEF-Tuを認識できるようになったことを明らかにした[95]。また、2015年にはコムギにシロイヌナズナのEFRを導入し、elf18に対する応答や、それに伴って起こる免疫マーカー遺伝子の発現誘導やカロースの沈着を確認し、実際に病原菌であるPseudomonas syringae pv. oryzaeに感染させた際に病変サイズや細菌の増殖が抑制されることを確認したとする論文が発表された[96]。
FLS2もEFRも同じようなシグナル伝達経路でPTIを開始させる。シロイヌナズナは、植物における免疫反応の制御を理解するために、これらのシグナル伝達経路の解明に貢献してきた。最も顕著なものとしては、MAPキナーゼカスケードがある。PTIの下流で起こる反応には、カロースの沈着、酸化バースト、防御関連遺伝子の転写が含まれる[97]。また、 FLS2およびEF-Tu受容体を持つ植物は、集団内の適応度が高いことが示されている[98]。このことから、植物-病原体抵抗性は、捕食の増加や異常気温などの動的環境に対応するため、世代を超えて蓄積された進化の仕組みであるという考え方がある[98]。
PTIは、非特異的な方法で病原体に対抗することができる。これは、病原体が分泌し、宿主の機能を変化させるエフェクタータンパク質を、抵抗性遺伝子(R遺伝子)が認識することに依存しており、しばしば遺伝子対遺伝子の関係として表現される。この認識は、直接的に行われる場合と、guardeeタンパク質を介して間接的に行われる場合があり、後者はガードモデルと呼ばれる。シロイヌナズナにおいて最初にクローニングされたR遺伝子はRPS2(resistance to Pseudomonas syringae 2)で、これはエフェクターであるavrRpt2を認識する役割がある[99]。エフェクタータンパク質であるavrRpt2はP. syringae pv. tomato strain DC3000のIII型分泌装置を介してシロイヌナズナに送り込まれる。RPS2によるavrRpt2の認識は、guardeeタンパク質であるRIN4を介して行われ、RIN4は切断される[100]。このとき、病原菌のエフェクターがRPS2に認識されると、過敏感反応と呼ばれる劇的な免疫反応が起こり、感染した植物細胞は細胞死を起こすことで、病原菌の拡散を防ぐ[82][101]。
植物における全身獲得抵抗性(Systemic Acquired Resistance、SAR)は、シロイヌナズナを使った研究により、理解が深まった抵抗性のもう一つの例である。サリチル酸の類縁物質であるベンゾチアジアゾール(BTH)は、作物の育成において抗真菌化合物として歴史的に使用されてきた化合物であり、サリチル酸と同様に植物にSARを誘導することが示されている[102]。SAR経路の起点となる反応は、シロイヌナズナで初めて示された。サリチル酸濃度の上昇を、細胞質での酸化還元変化によりnonexpresser of PR genes 1(NPR1)[103]が認識し、結果としてNPR1が還元される。NPR1は通常、オリゴマーとして存在するが、還元されるとモノマーになる[104]。モノマーになると核に移行し、多くのTGA転写因子と相互作用し、PR1などの病原菌関連遺伝子を誘導することができるようになる[105]。SARのもう一つの例として、細菌のサリチル酸水酸化酵素であるnahG遺伝子を発現する遺伝子組み換えタバコ植物で行われた研究では、その発現にサリチル酸の蓄積を必要とすることが示された[98]。
細胞内輸送は、病原体粒子を取り込んだり、あるいは取り込ませたりすることで、直接免疫に影響を与えるものではない。例えば、ダイナミン関連タンパク質をコードした2b/drp2b遺伝子は、侵入した物質を細胞内に移動させるのに役立ち、一部の変異体ではPstDC3000の病原性をさらに高めている[106]。
その他の研究
シロイヌナズナの組織を半in vitro状態で培養できるプラントオンチップ装置が報告されている[107]。この装置は、シロイヌナズナの花粉管誘導と有性生殖のメカニズムの理解に役立つと考えられている。
自家受粉
シロイヌナズナは主に自家受粉をする植物で、交配率は0.3%未満と推定されている[108]。一方、同じくシロイヌナズナ属に属するハクサンハタザオなどの近縁種は自家不和合性を持つため、自殖することができない。例えばハクサンハタザオでは、自身の花粉が柱頭についても花粉管を伸長させることができないため、種子を作ることができない[109][110]。そこで、シロイヌナズナにおいてゲノム全体の連鎖不平衡を解析した結果、自家受粉はおよそ100万年以上前に獲得した形質であることが示唆された[111]。自家受粉を引き起こす減数分裂は、遺伝的に大きな変異をもたらすことはないと考えられるが、こういった減数分裂は、各世代で生殖細胞が形成される際に、DNA損傷の組換え修復という適応的な利益をもたらすことができる[112]。このような利点は、自家受粉が行われても減数分裂が長期的に持続するのに十分であったかもしれない。シロイヌナズナにおける自家受粉の物理的なメカニズムは、開花前の自家受精であり、多くの場合受精は花が開く前に行われる。
データベースと情報源
ギャラリー

 花の拡大
花の拡大
 越冬時の葉の状態
越冬時の葉の状態
注釈
- かつてはAgrobacterium tumefaciensと呼ばれた
出典
- “Brassicaceae species checklist and database”. Species 2000 & ITIS Catalogue of Life. (2016). ISSN 2405-8858. オリジナルの9 December 2018時点におけるアーカイブ。 2016年6月1日閲覧。.
- NPO法人 自然観察大学『子どもと一緒に見つける 草花さんぽ図鑑』永岡書店、2019年、20頁。
- "Arabidopsis thaliana (L.) Heynh". Germplasm Resources Information Network (GRIN). Agricultural Research Service (ARS), United States Department of Agriculture (USDA). 2022年10月17日閲覧。
- Hoffmann, Matthias H. (2002). “Biogeography of Arabidopsis thaliana (L.) Heynh. (Brassicaceae)”. Journal of Biogeography 29: 125–134. doi:10.1046/j.1365-2699.2002.00647.x.
- Mitchell-Olds, Thomas (December 2001). “Arabidopsis thaliana and its wild relatives: a model system for ecology and evolution”. Trends in Ecology & Evolution 16 (12): 693–700. doi:10.1016/s0169-5347(01)02291-1.
- Sharbel, Timothy F.; Haubold, Bernhard; Mitchell-Olds, Thomas (2000). “Genetic isolation by distance in Arabidopsis thaliana: biogeography and postglacial colonization of Europe”. Molecular Ecology 9 (12): 2109–2118. doi:10.1046/j.1365-294x.2000.01122.x. PMID 11123622.
- “Planting molecular functions in an ecological context with Arabidopsis thaliana”. eLife 4: –06100. (March 2015). doi:10.7554/eLife.06100. PMC 4373673. PMID 25807084.
- “Arabidopsis thaliana”. Proceedings of the National Academy of Sciences of the United States of America 114 (20): 5213–5218. (May 2017). doi:10.1073/pnas.1616736114. PMC 5441814. PMID 28473417.
- “Arabidopsis thaliana (L.) Heynh.” (英語). www.gbif.org. 2019年6月1日時点のオリジナルよりアーカイブ。2018年12月8日閲覧。
- Hedberg, Olov (1957). “Afroalpine Vascular Plants: A Taxonomic Revision”. Acta Universitatis Upsaliensis: Symbolae Botanicae Upsalienses 15 (1): 1–144.
- “Archaic lineages broaden our view on the history of Arabidopsis thaliana”. The New Phytologist 219 (4): 1194–1198. (September 2018). doi:10.1111/nph.15244. PMID 29862511.
- “Arabidopsis thaliana – Overview”. Encyclopedia of Life. 2016年6月10日時点のオリジナルよりアーカイブ。2016年5月31日閲覧。
- “The rate and potential relevance of new mutations in a colonizing plant lineage”. PLOS Genetics 14 (2): e1007155. (February 2018). doi:10.1371/journal.pgen.1007155. PMC 5825158. PMID 29432421.
- “Arabidopsis thaliana (thale cress)”. Kew Gardens. 2018年2月28日時点のオリジナルよりアーカイブ。2018年2月27日閲覧。
- “State and Federal Noxious Weeds List | USDA PLANTS”. plants.sc.egov.usda.gov. 2018年12月9日時点のオリジナルよりアーカイブ。2018年12月8日閲覧。
- “IRMNG”. Encyclopedia of Life. 2018年4月1日時点のオリジナルよりアーカイブ。2022年10月17日閲覧。
- Flora of NW Europe: Arabidopsis thaliana Archived 8 December 2007 at the Wayback Machine.
- Blamey, M. & Grey-Wilson, C. (1989). Flora of Britain and Northern Europe. ISBN 0-340-40170-2
- Flora of Pakistan: Arabidopsis thaliana Archived 18 June 2008 at the Wayback Machine.
- Flora of China: Arabidopsis thaliana Archived 5 October 2018 at the Wayback Machine.
- “Flower Visitors in a Natural Population of Arabidopsis thaliana”. Plant Biology 5 (5): 491–494. (September 2003). doi:10.1055/s-2003-44784.
- “Bacillus megaterium rhizobacteria promote growth and alter root-system architecture through an auxin- and ethylene-independent signaling mechanism in Arabidopsis thaliana”. Molecular Plant-Microbe Interactions 20 (2): 207–17. (February 2007). doi:10.1094/MPMI-20-2-0207. PMID 17313171.
- “Arabidopsis thaliana: a model plant for genome analysis”. Science 282 (5389): 662, 679–82. (October 1998). Bibcode: 1998Sci...282..662M. doi:10.1126/science.282.5389.662. PMID 9784120.
- The 1001 Genomes Consortium (July 2016). “1,135 Genomes Reveal the Global Pattern of Polymorphism in Arabidopsis thaliana”. Cell 166 (2): 481–491. doi:10.1016/j.cell.2016.05.063. PMC 4949382. PMID 27293186.
- Archived 22 October 2016 at the Wayback Machine. TAIR: About Arabidopsis
- “Arabidopsis to rice. Applying knowledge from a weed to enhance our understanding of a crop species”. Plant Physiology 135 (2): 622–9. (June 2004). doi:10.1104/pp.104.040170. PMC 514098. PMID 15208410.
- “Complex life cycles of multicellular eukaryotes: new approaches based on the use of model organisms”. Gene 406 (1–2): 152–70. (December 2007). doi:10.1016/j.gene.2007.07.025. PMID 17870254. オリジナルの9 July 2021時点におけるアーカイブ。 2021年6月29日閲覧。.
- Novembre, John, ed (February 2010). “The scale of population structure in Arabidopsis thaliana”. PLOS Genetics 6 (2): e1000843. doi:10.1371/journal.pgen.1000843. PMC 2820523. PMID 20169178.
- “The protein encoded by the Arabidopsis homeotic gene agamous resembles transcription factors”. Nature 346 (6279): 35–9. (July 1990). Bibcode: 1990Natur.346...35Y. doi:10.1038/346035a0. PMID 1973265.
- “Röntgenmutationen bei Arabidopsis Thaliana (L) Heynh”. Naturwissenschaften 34: 26–28. (1947). doi:10.1104/pp.125.1.15.
- “Prehistory and history of Arabidopsis research”. Plant Physiology 125 (1): 15–9. (January 2001). doi:10.1007/BF00633319. PMC 1539315. PMID 11154286.
- “About AIS”. The Arabidopsis Information Resource (2018年11月8日). 2021年4月27日時点のオリジナルよりアーカイブ。2021年4月25日閲覧。
- “Transformation of Arabidopsis thaliana with Agrobacterium tumefaciens”. Science 234 (4775): 464–6. (October 1986). Bibcode: 1986Sci...234..464L. doi:10.1126/science.234.4775.464. PMID 17792019.
- “Molecular cloning and DNA sequence of the Arabidopsis thaliana alcohol dehydrogenase gene”. Proceedings of the National Academy of Sciences of the United States of America 83 (5): 1408–12. (March 1986). Bibcode: 1986PNAS...83.1408C. doi:10.1073/pnas.83.5.1408. PMC 323085. PMID 2937058.
- “Arabidopsis thaliana chloroplast, complete genome — NCBI accession number NC_000932.1”. National Center for Biotechnology Information. 2018年11月4日時点のオリジナルよりアーカイブ。2018年11月4日閲覧。
- “Complete structure of the chloroplast genome of Arabidopsis thaliana” (英語). DNA Research 6 (5): 283–290. (1999). doi:10.1093/dnares/6.5.283. ISSN 1340-2838. PMID 10574454.
- “Analysis of the genome sequence of the flowering plant Arabidopsis thaliana”. Nature 408 (6814): 796–815. (December 2000). doi:10.1038/35048692. PMID 11130711.
- “Comparisons with Caenorhabditis (approximately 100 Mb) and Drosophila (approximately 175 Mb) using flow cytometry show genome size in Arabidopsis to be approximately 157 Mb, thus approximately 25% larger than the Arabidopsis genome initiative estimate of approximately 125 Mb”. Annals of Botany 91 (5): 547–57. (April 2003). doi:10.1093/aob/mcg057. PMC 4242247. PMID 12646499.
- “Genome Assembly”. The Arabidopsis Information Resource. 2021年3月7日時点のオリジナルよりアーカイブ。2016年3月29日閲覧。
- “Evolution of genome size and chromosome number in the carnivorous plant genus Genlisea (Lentibulariaceae), with a new estimate of the minimum genome size in angiosperms”. Annals of Botany 114 (8): 1651–63. (December 2014). doi:10.1093/aob/mcu189. PMC 4649684. PMID 25274549.
- “Details - Arabidopsis_thaliana - Ensembl Genomes 63” (英語). ensembl.gramene.org. 2021年6月24日時点のオリジナルよりアーカイブ。2021年6月15日閲覧。
- “Arabidopsis thaliana (Mouse-ear cress)” (英語). www.uniprot.org. 2021年5月21日時点のオリジナルよりアーカイブ。2021年6月15日閲覧。
- Cheng, Chia-Yi; Krishnakumar, Vivek; Chan, Agnes P.; Thibaud-Nissen, Françoise; Schobel, Seth; Town, Christopher D. (2017). “Araport11: a complete reannotation of the Arabidopsis thaliana reference genome” (英語). The Plant Journal 89 (4): 789–804. doi:10.1111/tpj.13415. ISSN 1365-313X. PMID 27862469. オリジナルの24 June 2021時点におけるアーカイブ。 2021年6月15日閲覧。.
- “Arabidopsis thaliana ecotype Col-0 mitochondrion, complete genome — NCBI accession number BK010421”. National Center for Biotechnology Information (2018年10月10日). 2019年4月12日時点のオリジナルよりアーカイブ。2019年4月10日閲覧。
- “Physical mapping of the mitochondrial genome of Arabidopsis thaliana by cosmid and YAC clones”. Plant Journal 6 (3): 447–455. (1994). doi:10.1046/j.1365-313X.1994.06030447.x. PMID 7920724.
- “The plant mitochondrial genome: dynamics and maintenance”. Biochimie 100: 107–120. (2014). doi:10.1016/j.biochi.2013.09.016. PMID 24075874.
- “Floral dip: a simplified method for Agrobacterium-mediated transformation of Arabidopsis thaliana”. The Plant Journal 16 (6): 735–43. (December 1998). doi:10.1046/j.1365-313x.1998.00343.x. PMID 10069079.
- “Agrobacterium-mediated transformation of Arabidopsis thaliana using the floral dip method”. Nature Protocols 1 (2): 641–6. (2006). doi:10.1038/nprot.2006.97. PMID 17406292.
- “T-DNA Express: Arabidopsis Gene Mapping Tool”. signal.salk.edu. 2009年11月25日時点のオリジナルよりアーカイブ。2009年10月19日閲覧。
- “Eurasian Arabidopsis Stock Centre (uNASC)”. arabidopsis.info. 2001年12月12日時点のオリジナルよりアーカイブ。2009年10月19日閲覧。
- “New Arabidopsis recombinant inbred lines (Landsberg erecta x Nossen) reveal natural variation in phytochrome-mediated responses”. Plant Physiology 138 (2): 1126–35. (June 2005). doi:10.1104/pp.104.059071. PMC 1150426. PMID 15908601.
- “ABRC”. abrc.osu.edu. 2021年2月25日時点のオリジナルよりアーカイブ。2020年12月12日閲覧。
- “NASC Collection Info”. arabidopsis.info. 2011年7月19日時点のオリジナルよりアーカイブ。2011年2月15日閲覧。
- “Arabidopsis thaliana” (英語). Frontiers in Plant Science 8: 39. (2017). doi:10.3389/fpls.2017.00039. PMC 5258748. PMID 28174584.
- “Arabidopsis With Cas9-Based Gene Targeting”. Frontiers in Plant Science 9: 424. (5 April 2018). doi:10.3389/fpls.2018.00424. PMC 5895730. PMID 29675030.
- “Genome-wide non-mendelian inheritance of extra-genomic information in Arabidopsis”. Nature 434 (7032): 505–9. (March 2005). Bibcode: 2005Natur.434..505L. doi:10.1038/nature03380. PMID 15785770.Washington Post summary. Archived 18 November 2016 at the Wayback Machine.
- “Plant genetics: increased outcrossing in hothead mutants”. Nature 443 (7110): E8; discussion E8–9. (September 2006). Bibcode: 2006Natur.443E...8P. doi:10.1038/nature05251. PMID 17006468.
- “Genetics. Pollen contamination may explain controversial inheritance”. Science 313 (5795): 1864. (September 2006). doi:10.1126/science.313.5795.1864. PMID 17008492.
- Raikhel, Natasha V. (2017-04-28). “Firmly Planted, Always Moving”. Annual Review of Plant Biology (Annual Reviews (publisher)) 68 (1): 1–27. doi:10.1146/annurev-arplant-042916-040829. ISSN 1543-5008. PMID 27860488.
- “The mechanisms of UV mutagenesis” (英語). Journal of Radiation Research 52 (2): 115-125. (March 2011). doi:10.1269/jrr.10175. PMID 21436607.
- Kaiser G, Kleiner O, Beisswenger C, Batschauer A. Increased DNA repair in Arabidopsis plants overexpressing CPD photolyase. Planta. 2009 Aug;230(3):505-15. doi: 10.1007/s00425-009-0962-y. Epub 2009 Jun 12. PMID 19521716
- “First species of plant to flower in space” (英語). Guinness World Records 2017年3月10日閲覧。
- Letzter, Rafi (2019年1月4日). “There Are Plants and Animals on the Moon Now (Because of China)”. Space.com. 2019年1月15日時点のオリジナルよりアーカイブ。2019年1月15日閲覧。
- Keeter, Bill (2022年5月12日). “Scientists Grow Plants in Lunar Soil”. NASA. 2022年5月13日閲覧。
- Keeter, Bill (2022年5月12日). “Scientists Grow Plants in Lunar Soil”. NASA. 2022年5月14日閲覧。
- “Seed-to-Seed-to-Seed Growth and Development of Arabidopsis in Microgravity”. Astrobiology 14 (10): 866–875. (2014). Bibcode: 2014AsBio..14..866L. doi:10.1089/ast.2014.1184. PMC 4201294. PMID 25317938.
- “Lunar plant biology--a review of the Apollo era”. Astrobiology 10 (3): 261–74. (April 2010). Bibcode: 2010AsBio..10..261F. doi:10.1089/ast.2009.0417. PMID 20446867.
- “The war of the whorls: genetic interactions controlling flower development”. Nature 353 (6339): 31–7. (September 1991). Bibcode: 1991Natur.353...31C. doi:10.1038/353031a0. PMID 1715520.
- “Leaf development”. The Arabidopsis Book 11: e0163. (2013-06-07). doi:10.1199/tab.0163. PMC 3711357. PMID 23864837.
- “Vascular patterning”. The Arabidopsis Book 2: e0073. (2003-03-22). doi:10.1199/tab.0073. PMC 3243335. PMID 22303224.
- “Morphogenesis of simple and compound leaves: a critical review”. The Plant Cell 22 (4): 1019–32. (April 2010). doi:10.1105/tpc.109.073601. PMC 2879760. PMID 20435903.
- Moreno N, Bougourd S, Haseloff J and Fiejo JA. 2006. Chapter 44: Imaging Plant Cells. In: Pawley JB (Editor). Handbook of Biological Confocal Microscopy - 3rd edition. SpringerScience+Business Media, New York. p769-787
- “Imaging the live plant cell”. The Plant Journal 45 (4): 573–98. (February 2006). doi:10.1111/j.1365-313X.2006.02653.x. PMID 16441350.
- Simpson, Gordon G.; Dean, Caroline (2002-04-12). “Arabidopsis, the Rosetta Stone of Flowering Time?”. Science (American Association for the Advancement of Science (AAAS)) 296 (5566): 285–289. Bibcode: 2002Sci...296..285S. doi:10.1126/science.296.5566.285. ISSN 0036-8075. PMID 11951029.
- Friedman, Jannice (2020-11-02). “The Evolution of Annual and Perennial Plant Life Histories: Ecological Correlates and Genetic Mechanisms”. Annual Review of Ecology, Evolution, and Systematics (Annual Reviews (publisher)) 51 (1): 461–481. doi:10.1146/annurev-ecolsys-110218-024638. ISSN 1543-592X.
- Whittaker, Charles; Dean, Caroline (2017-10-06). “The FLC Locus: A Platform for Discoveries in Epigenetics and Adaptation”. Annual Review of Cell and Developmental Biology (Annual Reviews (publisher)) 33 (1): 555–575. doi:10.1146/annurev-cellbio-100616-060546. ISSN 1081-0706. PMID 28693387.
- “From seed to seed: the role of photoreceptors in Arabidopsis development”. Developmental Biology 260 (2): 289–97. (August 2003). doi:10.1016/S0012-1606(03)00212-4. PMID 12921732.
- “Circadian clock signaling in Arabidopsis thaliana: from gene expression to physiology and development”. The International Journal of Developmental Biology 49 (5–6): 491–500. (2005). doi:10.1387/ijdb.041968pm. PMID 16096959.
- “Arabidopsis plants perform arithmetic division to prevent starvation at night”. eLife 2: e00669. (June 2013). arXiv:1306.5148. doi:10.7554/eLife.00669. PMC 3691572. PMID 23805380.
- “Red-light-induced positive phototropism in Arabidopsis roots”. Planta 212 (3): 424–30. (February 2001). doi:10.1007/s004250000410. PMID 11289607.
- "Plants that Glow in the Dark" Archived 3 February 2014 at the Wayback Machine., Bioresearch Online, 18 May 2000
- “Bacterial disease resistance in Arabidopsis through flagellin perception”. Nature 428 (6984): 764–7. (April 2004). Bibcode: 2004Natur.428..764Z. doi:10.1038/nature02485. PMID 15085136.
- Hassani, M.A., Durán, P. and Hacquard, S. (2018) "Microbial interactions within the plant holobiont". Microbiome, 6(1): 58. doi:10.1186/s40168-018-0445-0.
 Material was copied from this source, which is available under a Creative Commons Attribution 4.0 International License Archived 16 October 2017 at the Wayback Machine.
Material was copied from this source, which is available under a Creative Commons Attribution 4.0 International License Archived 16 October 2017 at the Wayback Machine. - “SNARE-protein-mediated disease resistance at the plant cell wall”. Nature 425 (6961): 973–7. (October 2003). Bibcode: 2003Natur.425..973C. doi:10.1038/nature02076. PMID 14586469.
- “Pre- and postinvasion defenses both contribute to nonhost resistance in Arabidopsis”. Science 310 (5751): 1180–3. (November 2005). Bibcode: 2005Sci...310.1180L. doi:10.1126/science.1119409. hdl:11858/00-001M-0000-0012-3A32-0. PMID 16293760. オリジナルの11 March 2022時点におけるアーカイブ。 2019年9月5日閲覧。.
- “Arabidopsis PEN3/PDR8, an ATP binding cassette transporter, contributes to nonhost resistance to inappropriate pathogens that enter by direct penetration”. The Plant Cell 18 (3): 731–46. (March 2006). doi:10.1105/tpc.105.038372. PMC 1383646. PMID 16473969.
- “From perception to activation: the molecular-genetic and biochemical landscape of disease resistance signaling in plants”. The Arabidopsis Book 8: e012. (March 2010). doi:10.1199/tab.0124. PMC 3244959. PMID 22303251.
- “A single locus determines sensitivity to bacterial flagellin in Arabidopsis thaliana”. The Plant Journal 18 (3): 277–84. (May 1999). doi:10.1046/j.1365-313X.1999.00451.x. PMID 10377993.
- “FLS2: an LRR receptor-like kinase involved in the perception of the bacterial elicitor flagellin in Arabidopsis”. Molecular Cell 5 (6): 1003–11. (June 2000). doi:10.1016/S1097-2765(00)80265-8. PMID 10911994.
- Chinchilla D, Zipfel C, Robatzek S, Kemmerling B, Nürnberger T, Jones JD, Felix G, Boller T (2007). “A flagellin-induced complex of the receptor FLS2 and BAK1 initiates plant defence”. Nature 448: 479-500. PMID 17625569.
- Christopher Dardick, Pamela Ronald (2006). “Plant and animal pathogen recognition receptors signal through non-RD kinases”. PLoS Pathogens. PMID 16424920.
- “Transgeneration memory of stress in plants”. Nature 31 (442): 1046–49. (Aug 2006). doi:10.1038/nature05022. PMID 16892047.
- Urban, L.; Chabane Sari, D.; Orsal, B.; Lopes, M.; Miranda, R.; Aarrouf, J. (2018). “UV-C light and pulsed light as alternatives to chemical and biological elicitors for stimulating plant natural defenses against fungal diseases”. Scientia Horticulturae (Elsevier) 235: 452–459. doi:10.1016/j.scienta.2018.02.057. ISSN 0304-4238.
- “Perception of the bacterial PAMP EF-Tu by the receptor EFR restricts Agrobacterium-mediated transformation”. Cell 125 (4): 749–60. (May 2006). doi:10.1016/j.cell.2006.03.037. PMID 16713565.
- “Interfamily transfer of a plant pattern-recognition receptor confers broad-spectrum bacterial resistance”. Nature Biotechnology 28 (4): 365–9. (April 2010). doi:10.1038/nbt.1613. PMID 20231819.
- “Arabidopsis EF-Tu receptor enhances bacterial disease resistance in transgenic wheat”. New Phytologist 206 (2): 606-613. (April 2015). doi:10.1111/nph.13356. PMID 25760815.
- “Plant immunity triggered by microbial molecular signatures”. Molecular Plant 3 (5): 783–93. (September 2010). doi:10.1093/mp/ssq035. PMID 20713980.
- “A central role of salicylic Acid in plant disease resistance”. Science 266 (5188): 1247–50. (November 1994). Bibcode: 1994Sci...266.1247D. doi:10.1126/science.266.5188.1247. PMID 17810266.
- “RPS2, an Arabidopsis disease resistance locus specifying recognition of Pseudomonas syringae strains expressing the avirulence gene avrRpt2”. The Plant Cell 5 (8): 865–75. (August 1993). doi:10.1105/tpc.5.8.865. PMC 160322. PMID 8400869.
- “Arabidopsis RIN4 is a target of the type III virulence effector AvrRpt2 and modulates RPS2-mediated resistance”. Cell 112 (3): 379-89. (February 2003). doi:10.1016/s0092-8674(03)00040-0. PMID 12581527.
- “Initiation of RPS2-specified disease resistance in Arabidopsis is coupled to the AvrRpt2-directed elimination of RIN4”. Cell 112 (3): 369–77. (February 2003). doi:10.1016/S0092-8674(03)00036-9. PMID 12581526.
- “Benzothiadizaole induces disease resistance by a citation of the systemic acquired resistance signal transduction pathway”. The Plant Journal 10 (1): 71–82. (1996). doi:10.1046/j.1365-313x.1996.10010071.x. PMID 8758979.
- “Characterization of an Arabidopsis Mutant That Is Nonresponsive to Inducers of Systemic Acquired Resistance”. The Plant Cell 6 (11): 1583–1592. (November 1994). doi:10.1105/tpc.6.11.1583. PMC 160545. PMID 12244227.
- “Inducers of plant systemic acquired resistance regulate NPR1 function through redox changes”. Cell 113 (7): 935–44. (June 2003). doi:10.1016/S0092-8674(03)00429-X. PMID 12837250.
- “Salicylic acid and NPR1 induce the recruitment of trans-activating TGA factors to a defense gene promoter in Arabidopsis”. The Plant Cell 15 (8): 1846–58. (August 2003). doi:10.1105/tpc.012211. PMC 167174. PMID 12897257.
- Ben Khaled, Sara; Postma, Jelle; Robatzek, Silke (2015-08-04). “A Moving View: Subcellular Trafficking Processes in Pattern Recognition Receptor–Triggered Plant Immunity”. Annual Review of Phytopathology (Annual Reviews) 53 (1): 379–402. doi:10.1146/annurev-phyto-080614-120347. ISSN 0066-4286. PMID 26243727.
- Yetisen AK, Jiang L, Cooper JR, Qin Y, Palanivelu R, Zohar Y (May 2011). “A microsystem-based assay for studying pollen tube guidance in plant reproduction”. J. Micromech. Microeng. 25 (5): 054018. Bibcode: 2011JMiMi..21e4018Y. doi:10.1088/0960-1317/21/5/054018.
- “Population genetic structure and outcrossing rate of Arabidopsis thaliana (L.) Heynh”. Heredity 62 (3): 411–418. (1989). doi:10.1038/hdy.1989.56.
- Keita, Suwabe; Kaori, Nagasaka; Endang, Ayu Windari; Chihiro, Hoshiai; Takuma, Ota; Maho, Takada; Ai, Kitazumi; Hiromi, Masuko-Suzuki et al. (2006). Double-locking mechanism of self-compatibility in Arabidopsis thaliana: the synergistic effect of transcriptional depression and disruption of coding region in the male specificity gene. 23. pp. 1217–31. doi:10.3389/fpls.2020.576140.. PMID 33042191.
- 土松 隆志『植物はなぜ自家受精をするのか』慶應義塾大学出版会、2017年。ISBN 978-4-7664-2299-3。
- “The evolution of selfing in Arabidopsis thaliana”. Science 317 (5841): 1070–2. (August 2007). Bibcode: 2007Sci...317.1070T. doi:10.1126/science.1143153. PMID 17656687.
- Bernstein H; Byerly HC; Hopf FA; Michod RE (1985). "Genetic damage, mutation, and the evolution of sex". Science. 229 (4719): 1277–81. Bibcode:1985Sci...229.1277B. doi:10.1126/science.3898363. PMID 3898363
- “TAIR - Gene Expression - Microarray - Public Datasets”. 2021年12月4日時点のオリジナルよりアーカイブ。2021年12月4日閲覧。
外部リンク
 ウィキメディア・コモンズには、シロイヌナズナに関するカテゴリがあります。
ウィキメディア・コモンズには、シロイヌナズナに関するカテゴリがあります。- 植物遺伝子材料カタログ 理研 BRC 実験植物開発室
- シロイヌナズナ DNABookT
- シロイヌナズナ完全長cDNAクローン データ提供:理研ゲノム科学総合研究センター
- オックスフォード・ジャーナル(DNA Research)(英語)
- Arabidopsis transcriptional regulatory map
- The Arabidopsis Information Resource (TAIR)
- Salk Institute Genomic Analysis Laboratory
- What Makes Plants Grow? The Arabidopsis genome knows Featured article in Genome News Network
- The Arabidopsis book - A comprehensive review published yearly related to research in Arabidopsis
- A. thaliana protein abundance
- The Arabidopsis Information Portal (Araport)
_lehekarv_(trihhoom)_311_0804.JPG.webp)

